Resumen
Antecedentes
La hipertensión pulmonar (HP) se caracteriza por presiones elevadas en la arteria pulmonar debido a diversas causas, lo que afecta la función y la supervivencia del corazón derecho. La disulfidptosis, un mecanismo de muerte celular recientemente reconocido, puede desempeñar un papel en la HP, pero sus genes asociados (DiG) no se comprenden bien en este contexto. Este estudio tiene como objetivo definir la relevancia diagnóstica de los DiG en HP.
Métodos
Utilizando datos de GSE11726, analizamos los DiG y sus características inmunes para identificar genes centrales que influyen en la progresión de la PH. Se compararon varios modelos de aprendizaje automático, incluidos RF, SVM, GLM y XGB, para determinar cuál es el modelo de diagnóstico más eficaz. La validación utilizó los conjuntos de datos GSE57345 y GSE48166. Además, se estableció una red de CeRNA y se utilizó un modelo de rata con PH inducida por hipoxia para la validación experimental con análisis de transferencia Western.
Resultados
Se identificaron 12 DiG asociados significativamente con la HP. El modelo XGB destacó en precisión diagnóstica (AUC = 0,958), identificando los genes centrales DSTN, NDUFS1, RPN1, TLN1 y MYH10. Los conjuntos de datos de validación confirmaron la eficacia del modelo. Se construyó una red de CeRNA que involucra estos genes, 40 miRNA y 115 lncRNA. La predicción de fármacos sugirió un potencial terapéutico para el ácido fólico, respaldado por sólidos resultados de acoplamiento molecular. Validación experimental en un modelo de rata alineada con estos hallazgos.
Conclusión
Descubrimos los distintos patrones de expresión de DiG en PH, identificamos genes centrales utilizando un modelo de aprendizaje automático XGB y establecimos una red CeRNA. Se predijeron fármacos dirigidos a los genes centrales y se sometieron a acoplamiento molecular. También se realizó una validación experimental para estos genes centrales.
Introducción
La hipertensión pulmonar (HP) es una enfermedad progresiva causada por diversas etiologías y mecanismos patogénicos complejos, que conduce a alteraciones en la función pulmonar, aumento de la resistencia vascular pulmonar y elevación de la presión de la arteria pulmonar (1, 2). Estos cambios dan como resultado una disfunción del ventrículo derecho, que en última instancia causa insuficiencia cardíaca derecha y la muerte. Estudios epidemiológicos recientes han demostrado una tasa de incidencia de 97 por millón de adultos, que actualmente afecta a unos 100 millones de personas en todo el mundo. Las últimas directrices nacionales e internacionales para el diagnóstico de HP son las siguientes: una presión media de la arteria pulmonar superior a 20 mmHg en reposo o superior a 30 mmHg durante el ejercicio (3, 4). Debido a la compleja patogénesis de la HP y los síntomas tardíos, los pacientes a menudo se encuentran en una etapa moderada o avanzada cuando reciben el tratamiento clínico, momento en el que los tratamientos convencionales no pueden mejorar la afección de manera efectiva. Por tanto, es necesaria más investigación sobre los mecanismos del desarrollo de la HP.
Se ha informado que el desarrollo de la HP implica varios procesos de muerte celular programada que incluyen apoptosis, necrosis, piroptosis, ferroptosis y cuproptosis en las células del músculo liso de la arteria pulmonar.3, 5,6,7,8). Un tipo de muerte celular recientemente identificado, conocido como disulfidptosis, ha surgido como un programa de muerte celular distinto.9, 10). Este proceso se desencadena por un exceso de cistina, lo que provoca un estrés por disulfuro que conduce al colapso del citoesqueleto de actina. La proteína SLC7A11 es crucial para transportar cistina al interior de las células. Los niveles elevados de SLC7A11 provocan escasez de glucosa, lo que altera la producción de NADPH a través de la vía de las pentosas fosfato (10). Esta alteración da como resultado una acumulación excesiva de pequeñas moléculas de disulfuro, lo que desencadena una serie de cambios oxidativos que finalmente inducen la muerte celular.11). El papel de los genes relacionados con la disulfidptosis (DiG) en la HP sigue siendo poco comprendido y la mayoría de los estudios se concentran en sus implicaciones en el cáncer. Un estudio reciente indica que la aparición de muerte inducida por disulfiram está asociada con el desarrollo de enfermedad pulmonar obstructiva crónica (12). En la progresión de la HP, durante la proliferación del músculo liso de la arteria pulmonar, el aumento de la expresión de ROS altera la actividad transcripcional de las proteínas relevantes, induciendo la formación de enlaces disulfuro en las proteínas, lo que afecta aún más la función mitocondrial y el metabolismo celular, lo que lleva a la exacerbación de la HP. (13). Sin embargo, aún no se comprende completamente la función exacta de los DiG en el inicio y avance de la HP. Por lo tanto, explorar cómo los DiG contribuyen a la HP proporcionará una nueva perspectiva para el diagnóstico y tratamiento de la HP.
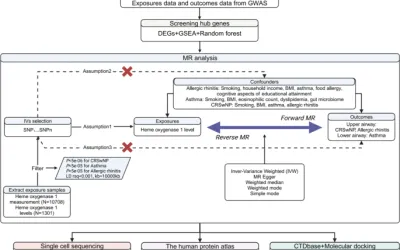
En esta investigación, analizamos datos de pacientes con HP de la base de datos Gene Expression Omnibus (GEO) (https://www.ncbi.nlm.nih.gov/geo/) para construir y evaluar un modelo de predicción para DiG. Este modelo tiene como objetivo evaluar con precisión la importancia diagnóstica de los DiG en personas con HP. Demostró una alta precisión predictiva con conjuntos de datos de entrenamiento y validación, y destacó un vínculo notable entre la infiltración inmune mediada por DiGs y la progresión de la HP. Además, la expresión de los objetivos centrales de DiG se validó adicionalmente utilizando un modelo de ratas con hipertensión pulmonar hipóxica (HPH) (Fig. 1).
Figura 1El diseño del estudio.
Imagen a tamaño completoMateriales y métodos
fuente de datos
GSE117261 (2), GSE24988 (14) y GSE33463 (15) los conjuntos de datos se obtuvieron de GEO (http://www.ncbi.nlm.nih.gov/geo/) base de datos (tabla T1) (16). Los datos de GSE117261 eran un conjunto de datos de entrenamiento (25 muestras de control y 58 de PH) (17). Se utilizaron GSE24988 y GSE33463 como conjuntos de datos de validación.
Selección de DiGs expresados diferencialmente.
Los DiG se seleccionaron recuperando 23 genes de un estudio anterior (9, 10). Se empleó una prueba t para examinar las diferencias entre los grupos de pacientes de control y HP. Los resultados, que ilustran variaciones en la expresión genética, se mostraron en mapas de calor (18, 19).
Análisis de conglomerados basado en DiG expresados diferencialmente
El análisis de conglomerados se realizó con ConsensusClusterPlus en el conjunto de entrenamiento utilizando DiGs (20). El conjunto de datos de entrenamiento se sometió a una evaluación mediante el Análisis de variación del conjunto de genes (GSVA) (21).
Construcción de modelos predictivos basados en múltiples métodos de aprendizaje automático.
Se utilizó un conjunto diverso de técnicas de aprendizaje automático para desarrollar modelos predictivos para diagnosticar la HP. Estos modelos incluían el modelo de aumento de gradiente extremo (XGB) (22), bosque aleatorio (RF) (23), modelo lineal generalizado (GLM) (24) y máquina de vectores de soporte (SVM) (25). La distribución de los residuos del modelo entre estos cuatro métodos se visualizó utilizando el paquete DALEX. Se eligió el modelo de aprendizaje automático más eficaz, identificando los cinco genes principales como marcadores de diagnóstico clave. El rendimiento del modelo se evaluó visualizando el área bajo la curva (AUC) de las curvas de característica operativa del receptor (ROC) con el paquete R ‘pROC’.
Construcción y validación de nomogramas.
Para validar y operacionalizar aún más la capacidad predictiva, se construyó un nomograma utilizando el paquete R «RMS». A cada predictor se le asignó una puntuación, y la «puntuación total» representa la suma acumulada de estas puntuaciones. Para evaluar la precisión predictiva del nomograma, se utilizaron curvas de calibración y análisis de curvas de decisión (DCA).
Análisis de la infiltración de células inmunes en PH.
El análisis de 22 células inmunes se realizó utilizando el algoritmo CIBERSORT (https://cibersort.stanford.edu/). Este método emplea el muestreo de Monte Carlo para derivar valores p para la deconvolución de cada muestra. La significación se estableció cuando los valores de p eran inferiores a 0,05 y la suma de las proporciones de los 22 tipos de células inmunes en cada muestra era igual a uno (26).
Construcción de la red CeRNA
La red lncRNA-miRNA-mRNA ceRNA se estableció prediciendo las interacciones miRNA-miRNA utilizando las bases de datos TargetScan, miRDB y miRanda. Las interacciones entre miARN y lncRNA se identificaron utilizando la base de datos SpongeScan. Luego, la red se construyó utilizando el software Cytoscape (versión 3.7.2).
Identificación de fármacos candidatos de molécula pequeña.
Los fármacos terapéuticos que se dirigen a genes centrales se identificaron en la base de datos CTD (http://ctdbase.org/). Luego se construyó una red de interacción fármaco-gen utilizando el software Cytoscape (versión 3.7.2). Además, el acoplamiento molecular de los fármacos candidatos con genes centrales se llevó a cabo utilizando el software Autodock Vina (versión 1.1.2). Los resultados de estas interacciones moleculares se visualizaron utilizando PyMOL (versión 2.0.0) (27).
Animal
Se compraron ratas SPF macho en el Centro de Animales de la Universidad Médica de Bengbu, provincia de Anhui (SYXK 20201104). Todos los procedimientos se adhirieron estrictamente a las «Directrices del Consejo Chino de Investigación Animal» y las «Directrices de cuidado animal» para minimizar el sufrimiento de los animales. Las ratas se alojaron en habitaciones bien ventiladas, con temperatura moderada (22 ± 2 °C), humedad adecuada (55 ± 5%), un ciclo de luz/oscuridad de 12 horas y libre acceso a comida y agua. Después de una semana de aclimatación con alimentación estándar para ratones de laboratorio, los ratones se dividieron aleatoriamente en un grupo de control (n = 6) y un grupo HPH (HPH, n = 6). El grupo HPH se colocó en una cámara hipóxica (que contenía cloruro de sodio anhidro y cal sodada para absorber agua y CO2), con libre acceso a alimentos y agua. Se introdujo N2 en la cámara hipóxica a una velocidad de 8 l/min, luego se ajustó a 1 l/min después de media hora para mantener la concentración de oxígeno al 10 % según lo monitoreado por un medidor de oxígeno. Las ratas fueron expuestas a hipoxia durante 8 h por día, durante 6 días consecutivos cada semana, con un día de descanso por semana, por un total de 4 semanas, lo que puede resultar en la formación de un modelo de rata HPH (28).
Productos químicos y materiales.
Pentobarbital sódico (Siasia Reagent, China), Anticuerpos: NDUFS1 (Abcam, ab169540, EE. UU.), RPN1 (Abcam, ab198508, EE. UU.), TLN1 (Abcam, ab108480, EE. UU.), DSTN (affinity, DF15268, China), MYH10 (Proteintech , 19673-1-AP, China), β-actina (CST, 4970, EE. UU.), IgG de cabra anti-conejo (conjugado HRP) (CST, 14708, EE. UU.) e IgG de cabra anti-ratón (conjugado HRP) (CST, 14709, EE. UU.).
Examen histopatológico del pulmón.
Los pulmones de las ratas se fijaron en formaldehído al 4% deshidratado con concentraciones variables de etanol, seguido de inclusión y corte en parafina, y…